version_name = "3_var_log"
hyp_reg_log = hypotheses_regression_lineaire(y, X, df, version_name, details=None)
| valeurs_reelles | valeurs_predites | residus | |
|---|---|---|---|
| 705507 | 5.004370 | 5.598458 | -0.594088 |
| 4248006 | 9.863347 | 7.925396 | 1.937951 |
| 491434 | 9.156045 | 8.991307 | 0.164739 |
| 1327638 | 9.662478 | 9.418913 | 0.243564 |
| 1367283 | 7.371434 | 7.645321 | -0.273887 |
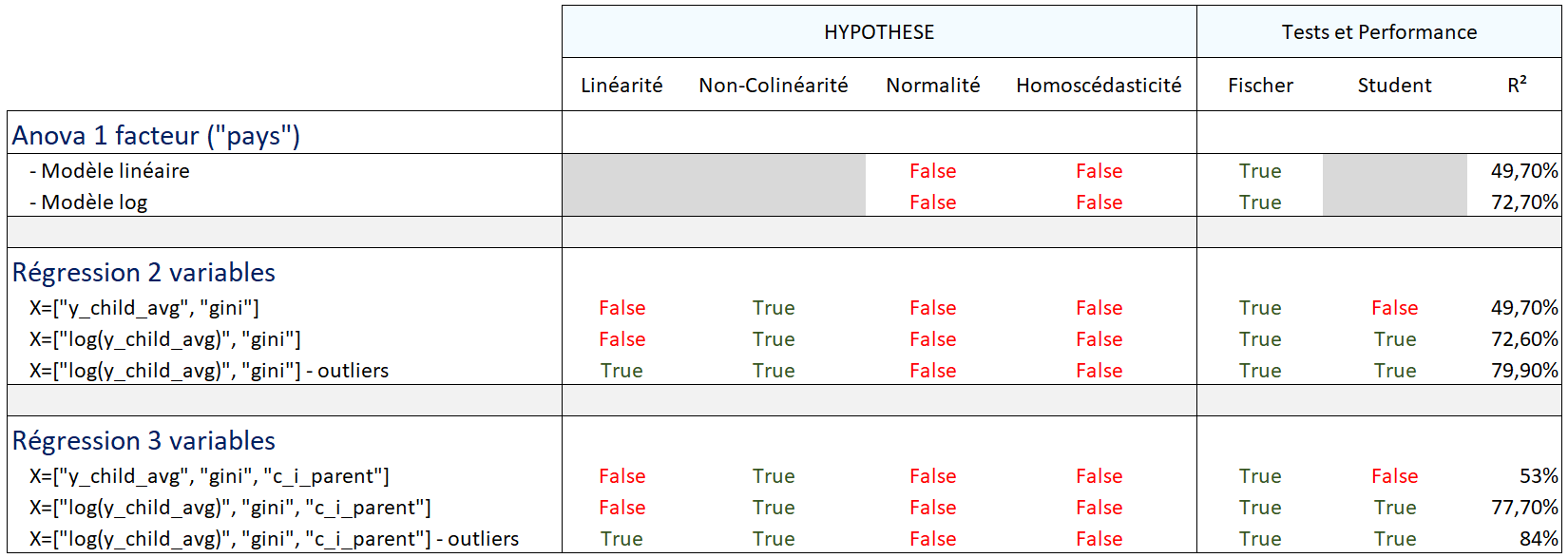
=> si linéarité, alors les points de la droite "prédictions / valeurs réelles" devraient suivre la droite d'équation y=x
| pearson | p-value | |
|---|---|---|
| gini__c_i_parent | 0.000015 | 0.971967 |
| ln_y_child_avg__c_i_parent | 0.000055 | 0.894622 |
| ln_y_child_avg__gini | -0.261327 | 0.000000 |
Toutes les |corrélations croisées| sont inférieures à 0.8 => absence de colinéarité entre les variables explicatives
R² de la régression : 0.781
Selon le critère de Klein : absence de colinéarité entre les variables explicatives (toutes les corrélations croisées au carré sont inférieures au R² de la régression)
| feature | VIF | |
|---|---|---|
| 0 | ln_y_child_avg | 1.073297 |
| 1 | gini | 1.073297 |
| 2 | c_i_parent | 1.000000 |
- H0 : Les résidus suivent une loi normale
- H1 : Les résidus ne suivent pas une loi normale
- Seuil de signification : 5%
Interprétation des tests :
- si p_value < 0.05 : on rejette H0 : les variables explicatives ne suivent pas une loi normale
- si p_value >= 0.05 : on accepte H0 : les variables explicatives suivent une loi normale
Jarque-Bera test ---- statistic: 1805609.6352, p-value: 0.0 Kolmogorov-Smirnov test ---- statistic: 0.1277, p-value: 0.0000
Les 2 tests conduisent à rejeter H0 : on en conclue que les données ne suivent pas une loi normale
Interprétation des tests :
- si p_value < 0.05 : on rejette H0 : on rejette l'hypothèse d'homoscédasticité : les variances ne sont pas constantes
- si p_value >= 0.05 : on accepte H0 : on accepte l'hypothèse d'homoscédasticité : les variances sont constantes
Breusch Pagan test ---- p-value: 0.0
La p_value est inférieure au seuil de signification de 5% : on rejette l'hypothèse H0 d'homoscédasticité des résidus => les variances ne sont pas constantes
- Hypothèse de linéarité : se référer au graphique de linéarité
- Absence de colinéarité entre les variables explicatives
- Non normalité des variables explicatives
- Hétéroscédasticité des résidus